
Produits
Découverte chimioprotéomique de nouvelles structures principales pour des cibles non médicamenteuses
Contexte technique
Actuellement, seulement environ 800 protéines ont été ciblées par des médicaments approuvés par la FDA, et un grand nombre de cibles liées à la maladie sont « inmédicamentables ». Car, à l’heure actuelle, la plupart des technologies reposent sur des protéines purifiées. L’avènement de la chimprotéomique a révolutionné la découverte de médicaments, depuis les protéines purifiées jusqu’aux cellules vivantes. Il est capable d’analyser quantitativement les interactions entre petites molécules et protéines à l’échelle du protéome humain. Aujourd'hui, la découverte de structures covalentes contre des résidus d'acides aminés spécifiques de protéines cibles, telles que la cystéine, la lysine, la méthionine et la tyrosine, a été démontrée dans les lysats cellulaires et les cellules vivantes. ChomiX exploitera sa propre plateforme chimioprotéomique pour franchir rapidement les barrières des cibles « non médicamentables ».

Avantages techniques
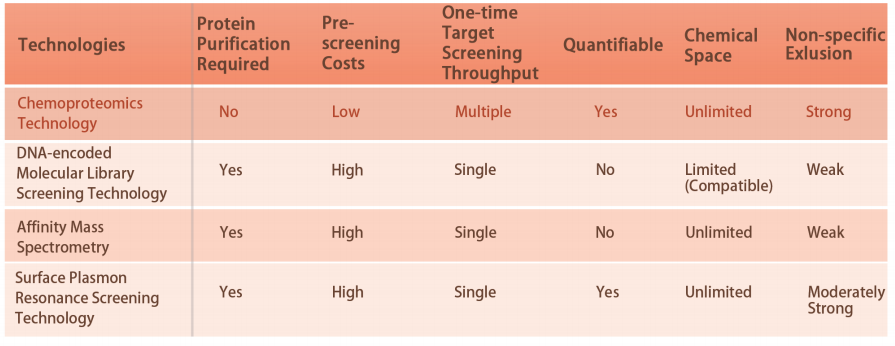
Caractéristiques techniques de la plateforme
La plateforme DIA-ABPP est conçue pour découvrir des liants covalents qui engagent des cibles protéiques via des réactions covalentes avec le groupe thiol des résidus cystéine. Une fois que les résidus cystéine actifs des protéines sont occupés préférentiellement par des molécules covalentes, le marqueur de la sonde universelle qui réagit spécifiquement avec le groupe thiol sera mis en compétition. En combinaison avec des techniques de protéomique quantitative basées sur DIA, les intensités des peptides marqués par une sonde entre la molécule covalente et le groupe témoin pourraient être quantifiées. Ces différences de signaux représentent les taux d'occupation des molécules covalentes contre leurs protéines de liaison. Contrairement aux stratégies traditionnelles de criblage à cible unique, la plateforme DIA-ABPP permet une analyse quantitative des interactions entre les fragments électrophiles et presque toutes les protéines des cellules vivantes avec la résolution des résidus d'acides aminés, ce qui améliore considérablement l'efficacité et réduit les risques pour le projet de découverte de structures principales à haut débit. .

Flux de travail de criblage de composés de plomb covalents
Caractéristique 1

Notre bibliothèque de molécules de type médicament contient une variété de groupes réactifs (ogives) caractérisés par une légère réactivité électrophile, tels que l'acrylamide, le chloroacétamide et l'alcynamide. Ces molécules comportent des pharmacophores structurellement divers qui sont conformes à la règle des cinq de Lipinski.
Caractéristique 2

Actuellement, la bibliothèque de cibles protéiques capturées par la sonde chimique spécifique du thiol couvre 39 962 sites de cystéine provenant de 12 421 protéines, notamment des kinases, des phosphatase, des ligases et des facteurs de transcription. La bibliothèque reste continuellement mise à jour, garantissant l'accès aux dernières informations sur des cibles et des sites spécifiques. Des informations détaillées concernant des cibles et des sites spécifiques sont disponibles via le lien ci-dessous.
Caractéristique 3

En une seule expérience de criblage, la plateforme est capable de quantifier l’engagement cible de petites molécules (A2-H6) contre toutes les protéines marquées dans l’ensemble du protéome. La figure ci-dessus montre l'occupation du site de cibles cliniquement importantes sélectionnées par des molécules covalentes. Plus la couleur est foncée, plus l'occupation des molécules dans les poches de liaison, y compris les sites orthostériques et allostériques, est élevée (l'astérisque indique que le taux d'occupation est supérieur à 80 %). Ainsi, l’affinité et la sélectivité de chaque molécule peuvent être évaluées simultanément pour une optimisation ultérieure de la structure.




